研究人员开发算法来识别低微生物生物量微生物群落中的微生物污染物
微生物组科学的主要挑战之一是将潜在的环境污染物与真正的、真正的微生物组信号区分开来。与低生物量环境下的宏基因组测序相关的挑战包括真实信号与污染、来自采样套件或提取套件或环境的残余DNA之间的区别。
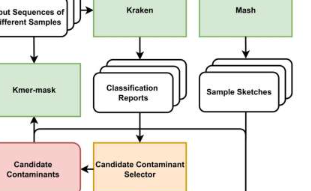
虽然研究人员通常包括来自设备或环境的阴性对照,并使用算法工具来识别环境中存在的分类群,但并非所有数据集都带有阴性对照。贝勒医学院和莱斯大学的研究人员开发了一种从头污染检测工具,以建立微生物鉴定和分析的可重复性。他们的发现最近发表在NatureCommunications上。
“我们与莱斯大学的合作者合作开发和测试了一种我们称为Squeegee的计算工具,”贝勒和德克萨斯儿童医院妇产科教授KjerstiAagaard博士说。
“Squeegee的前提是我们可以使用计算机分析管道来帮助我们检测污染物的‘面包屑’,这些污染物预计在所有人类(或其他哺乳动物)宿主和采样或实验室环境中发现的微生物组之间是常见的。”
贝勒的Aagaard实验室在过去十年中进行了研究,从大量参与者那里获得了大量丰富的数据集,这些数据集的生物量特别低,并且有许多阴性对照。他们与赖斯Treangen实验室的研究人员合作测试Squeegee,这是一种用于人类研究生命数据集的算法,该算法具有来自不同环境和DNA提取套件的污染控制。
他们研究了假阳性率、召回率以及Squeegee在没有阴性对照的情况下预测和标记这些环境污染组的准确性。
“我们能够证明Squeegee能够在这些地面实况数据集中具有高加权召回率和非常低的假阳性率,”贝勒妇产科博士后研究员MichaelJochum博士说。
根据Jochum的说法,Squeegee提高了低生物量研究中宏基因组测序分析结果的整体可靠性,这些研究包含少量微生物DNA,如母乳、胎盘或羊水。从头污染识别工具能够识别批次效应,并将其标记为潜在污染物。鉴于Aagaard实验室在研究这些稀疏微生物环境方面的重点和专业知识,他们已将此工具添加到他们的工具箱中,以用于正在进行和未来的研究。
声明:本站所有文章资源内容,如无特殊说明或标注,均为采集网络资源。如若本站内容侵犯了原著者的合法权益,可联系本站删除。










